
新基因功能数据可供其他科学家使用。图片来源:Jen Cook-Chrysos/Whitehead Institute
建立遗传变化和表型之间的关联对于理解基因和细胞功能至关重要。经典的研究方式主要包括以表型为中心的“正向遗传”,即揭示驱动表型的基因变化;以及以基因为中心的“反向遗传”,即对确定的遗传变化引起的不同表型进行解析。
近年来,基因技术的革新也推动了表观遗传遗传研究的进展。其中,CRISPR-Cas9基因编辑技术可以轻松地对基因进行编辑,进而抑制或激活基因,在揭示基本细胞机制、分化因子和遗传疾病相关基因以及识别癌症驱动基因等层面提供了有力工具。单细胞技术的发展也使在单细胞层面读取表观遗传学、转录组学、蛋白质组学和成像信息等成为可能,同时单细胞维度的研究也可以深入分析选择性遗传扰动影响的具体细胞类型和细胞状态。
因此,单细胞CRISPR筛选可以同时分析单细胞的遗传干扰和高维表型,从而将正向遗传学的基因与反向遗传学丰富的表型相结合。虽然单细胞CRISPR筛选技术前景广阔,但其应用仅限于最多几百个基因扰动研究,并且这些基因扰动研究也通常被用来解决预先确定的生物学问题。目前,高通量、无偏颇的单细胞CRISPR筛选研究仍然缺失。


文章发表在Cell
近日,美国加州大学旧金山分校与纪念斯隆·凯特琳癌症中心等单位的研究人团队合作Cell期刊发表了题为“Mapping information-rich genotype-phenotype landscapes with genome-scale Perturb-seq”的研究性文章。研究团队利用一种紧凑的、多路CRISPR干扰文库(CRISPRi),结合单细胞转录组测序、Perturb-seq技术等分析了数千个功能缺失的基因扰动在不同细胞类型中的作用,揭示了细胞表型、基因功能和调控网络的多维信息,绘制了第一个全面的人类细胞基因型-表型综合图谱。

研究概要图,来源:Cell
全基因组Perturb-seq的多路CRISPRi策略
Perturb-seq是指利用CRISPR-Cas9技术将基因变化引入细胞内,然后使用单细胞转录组测序捕获特定基因变化导致的转录组信息变化,能够研究给定细胞类型的全面遗传扰动影响,可以以前所未有的深度跟踪打开或关闭基因的影响。基于Perturb-seq,研究团队探究了可以提高可扩展性和数据质量的关键参数,例如遗传扰动模式和sgRNA库,并最终设计了一种包含多个时间点和细胞类型的Perturb-seq筛选方法,并可利用10x Genomics的液滴法单细胞转录组测序技术对所有筛选策略下的细胞状态进行解析。

图1. 基因组尺度Perturb-seq的多路CRISPRi策略示意图,来源:Cell
为了揭示基因扰动的功能后果和基因型-表型关系,研究团队使用人类血癌细胞系以及来自视网膜的非癌细胞,对超过250万个细胞进行了Perturb-seq,并使用这些数据构建了一个基因型-表型综合图谱。研究团队根据基因的共同调控将其聚类到特定表达程序中,并计算每个扰动簇中每个基因表达程序的平均活性。分析结果包含多个与基因干扰相关的已知表达程序,包括蛋白酶体功能障碍导致的蛋白酶体亚基上调、 ESCRT蛋白缺失时NF-kB信号通路的激活,以及胆固醇生物合成上调对囊泡运输缺陷的反应等。
有趣的是,聚类分析发现了许多驱动红系或髓系分化的基因扰动,与K562细胞的多系潜能也是一致的。正如预期的那样,红细胞生成的关键调控因子(GATA1、LDB1、LMO2和KDM1A)的缺失导致了髓系分化增强,BCR-ABL及其适配体GAB2的抑制则促进了红细胞的分化。
接下来,研究团队分析了选择性必需基因的分化作用,因为这些基因可能是颇具前景的治疗靶点。研究发现,在K562细胞中必需的酪氨酸磷酸酶PTPN1的缺失驱动了髓细胞分化。此外,在靶向实验中,联合敲除PTPN1和KDM1与单独敲除任意一个基因相比,导致分化和生长缺陷的表型会显著增加,表明这些靶点是通过不同的细胞机制发挥作用。以上结果强调了表型在了解细胞分化和治疗靶点方面的效用。

图2. 基于Perturb-seq的基因型与表型关系汇总,来源:Cell
探索单细胞异质性可以揭示在整体或平均检测中被遗漏的机制。为了评估基因扰动诱导表型的外显率,研究团队采用SVD评分作为单细胞表型大小的衡量标准,通过单细胞SVD分数的变化对基因扰动进行表型影响评估。SVD评分是量化每个受扰动细胞的转录组相对于对照细胞的离群程度。
分析结果表明,许多与染色体分离有关的基因都是细胞异质性的主要驱动因素,包括TTK、SPC25、DSN1,这些遗传干扰导致的极端转录变化可能是由于有丝分裂错误分离导致的染色体拷贝数的急性变化。
为了探究这一点,研究人员使用inferCNV估算了基因组中单细胞DNA拷贝数变异。与预期一致,干扰纺锤体装配检查点的核心组成部分TTK,可以导致非整倍体和近整倍体细胞的染色体拷贝数发生显著变化。此外,干扰TTK的细胞中有76%发生了核型改变,未受干扰的细胞中只有2%发生了核型改变。值得注意的是,由于染色体的随机增加或减少,TTK敲除细胞具有高度可变的核型,这也是其表型异质性的原因。同时,该分析还揭示了单细胞CRISPR筛选可以用来解析表型,而不是预先定义的实验终点。

图3. 单细胞中非整倍体的基因驱动和后果,来源:Cell
当前,领域内一个关键的科学问题是如何理解细胞核和线粒体基因组的表达来应对线粒体压力。该最新研究的实验设计为探究这一问题提供了可能。为了确定基因扰动引起的差异表达模式,研究团队检测了单细胞转录组测序数据在线粒体基因组中的分布。为了验证这种基于位置的分析的有效性,首先证实了已知线粒体转录调控因子(TEFM)和RNA降解(PNPT1) 的敲除会导致线粒体基因组位置发生重大变化。
相比之下,研究发现许多基因扰动似乎导致了mRNA相对丰度的变化,而不是位置排列的总体变化。鉴于观察到的反应的复杂性,研究人员提出可能有多种机制影响不同线粒体编码转录本的水平,以应对不同的压力。
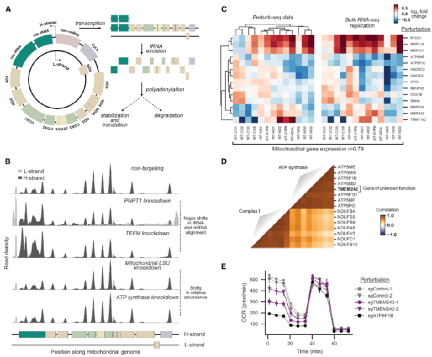

图4. 解析压力应激下线粒体基因组的调控机制,来源:Cell
单细胞CRISPR筛选代表了一种新兴的工具,可用于生成丰富的基因型-表现型图谱。但目前单细胞CRISPR筛选研究仅限于预先选择的基因,研究重点也是预先确定的生物学问题。在该最新研究中,研究团队进行了全基因组规模的单细胞CRISPR筛选,并展示了这些筛选策略是如何使用数据驱动的分析来解剖广泛的生物学现象,强调了关键的基因功能和衍生原则,同时绘制了丰富的基因型-表型图谱以指导未来的研究。该研究为系统探索遗传和细胞功能提供了源动力,同时也为领域提供了宝贵的数据资源。
在未来,研究人员希望将Perturb-seq用于癌细胞系之外的不同类型细胞研究,也希望继续探索基因功能图谱。文章共同通讯作者Thomas M. Norman博士表示:“该研究是多个科研团队多年合作工作的结晶,很高兴看到它继续取得成功和扩展,我认为这个数据集甚至将使来自生物医学以外领域的研究团队进行各种分析成为可能。”
参考文献:
1. Replogle et al., Mapping information-rich genotype-phenotype landscapes with genome-scale Perturb-seq, Cell (2022).
2. Fianu, I., et al., (2021). Structural basis of Integrator-mediated transcription regulation. Science 374, 883–887.
3. Kummer, E., and Ban, N. (2021). Mechanisms and regulation of protein synthesis in mitochondria. Nat. Rev. Mol. Cell Biol. 22, 307–325.
本文由 SEQ.CN 作者:白云 发表,转载请注明来源!






