近年来,空间转录组学(spatial transcriptomics)的迅速发展,对生物学的研究发展产生了重要影响。空间转录组可帮助科研工作者在不破坏组织原有空间形态的前提下,从分子水平了解生物组织之间复杂的交互网络关系,探索关键因素与环境之间的响应和变化。为科研工作者提供了研究复杂多细胞生物在组织系统和组织互作的工具,使得全面了解组织水平的基因表达图谱成为可能。
基于不同的空间转录组研究技术,科研工作者开发出五种不同研究策略(图1), 包括:1.基于显微分割基因表达的技术,2.原位杂交(ISH)技术,3.原位测序 (ISS)技术,4.原位捕获(ISC)技术,5.空间数据的计算模拟构建。
图1.空间转录组技术的简要历史时间表。来源:BioEssays[1]
目前,空间转录组研究的常用方法都存在局限性:基于原位测序(ISS)或杂交(ISH)的方法具有较高的分辨率和灵敏度,但通量有限,单次只能检测少数的基因,限制了其在探索全转录物相互作用时的通用性;基于原位捕获(ISC)的方法可以同时靶向所有带有poly(A)尾的转录本,但由于在组织水平的分辨率和灵敏度较低,对其应用和发展造成了限制。
为了克服当前实验方法的局限性,来自瑞典皇家理工学院(KTH)的Joakim Lundeberg教授研究团队提出了一种空间表达数据的深度生成模型,能够将ISC数据与高分辨率组织学图像结合,提升空间转录组的分辨率。近日,该研究团队将其研究成果发表在了Nature Biotechnology上,文章题为“Super-resolved spatial transcriptomics by deep data fusion”。
文章发表在Nature Biotechnology

文章通讯作者Joakim Lundeberg教授
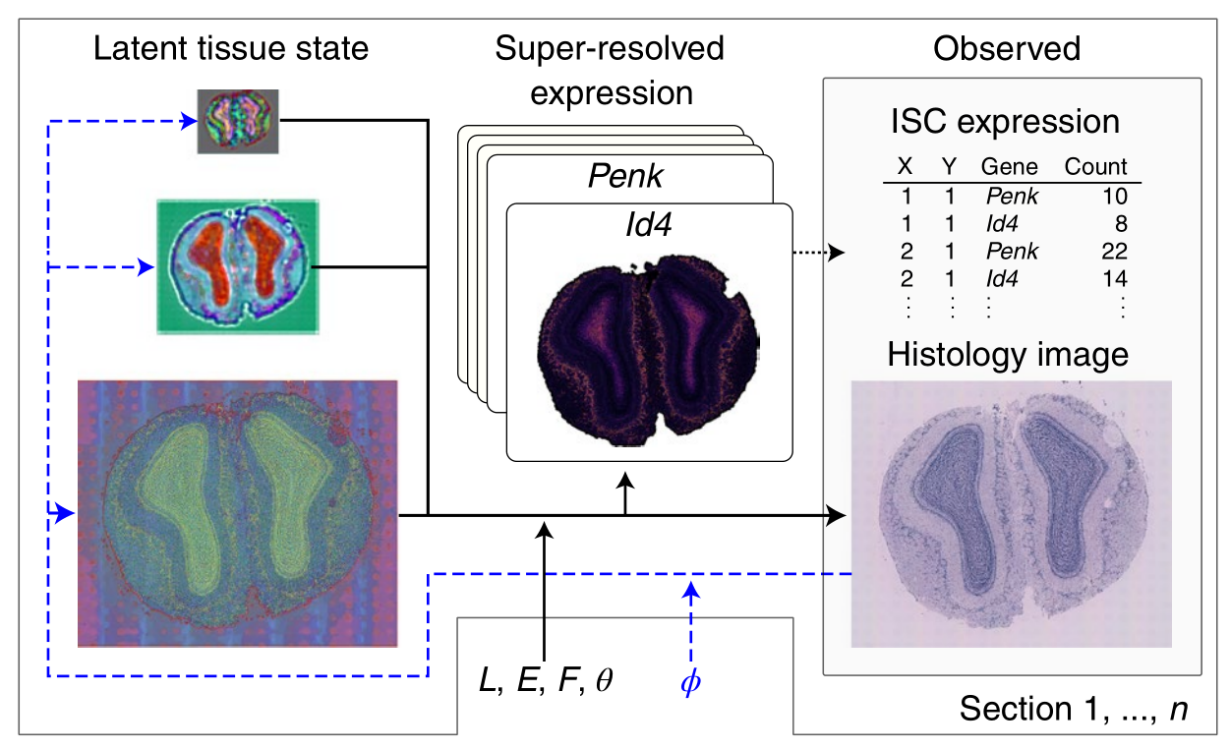
研究团队将ISC数据与高分辨率组织学图像进行深度融合,以推断转录组范围的超分辨率表达图。超分辨表达图量化了原始测量位置内和之间的基因表达(图2a)。此外,该模型还能够在联合实验中通过参考ISC切片数据,对来自同一组织的切片进行预测(图2b)。
图2.数据融合与预测示意图。来源:Nature Biotechnology[2]
基于建模分析,研究团队首先在多个空间分辨率上对组织学解剖图像进行了分析,充分捕获了全局以及局部的特征。通过神经网络进行学习和推理,将表达模式扩展到任意的解剖状态(图3) 。
图3.模型的建立示意图。来源:Nature Biotechnology[2]
研究人员在来自小鼠嗅球的12个组织切片数据中,验证了该模型的准确性。通过将推断的表达图与来自小鼠脑图谱的原位杂交数据进行比较,研究团队发现该模型推断的表达与参考数据相匹配(图4)。例如,Ntng1在二尖瓣细胞层(MCL)中的表达和Dusp14在MCL和颗粒层中的表达密切相关。相比之下,ISC数据较粗糙,无法解析相同的表达模式。
图4.模型的验证——小鼠嗅球的组织切片与表达量特征。来源:Nature Biotechnology[2]
此外,研究人员还使用该模型对小鼠嗅球和人类乳腺癌中微米级解剖特征的差异表达进行了研究(图5a、b)。研究人员首先分析了小鼠嗅球的MCL(图5c)并发现了几个明显上调和下调的基因(图5d)。接下来,研究人员通过分析肿瘤边缘和内部区域之间的转录组梯度(图5e)表征了细胞的组成类型。虽然癌细胞在DCIS病变中占主导地位,但研究人员仍观察到几个肿瘤相关基因在肿瘤边缘的50μm内被上调,测量之间的中心到中心距离的一半(图5f )。例如:C1QA、C1QB和C1QC等已被证明可促进血管生成和肿瘤生长;CD74则是乳腺癌转移性肿瘤生长的已知标志物,并且已被研究作为血癌中抗体-药物偶联疗法的潜在靶标。
图5.模型的应用——小鼠嗅球和人类乳腺癌解剖组织的基因差异表达。来源:Nature Biotechnology[2]
综上所述,该研究证明了组织学图像数据对组织中的空间表达模式具有高度的信息量,并提出了将ISC、高分辨率图像数据相融合的空间基因表达综合模型,可推断超分辨率表达图,能够用于研究详细解剖特征中的基因表达。此外,研究还证明了从组织学图像中预测空间基因表达的可行性,基于图像的计算机空间转录组学(ISST)将会在未来实现更广泛的研究与应用。
[1].Asp, M., Bergenstråhle, J., & Lundeberg, J. (2020). Spatially resolved transcriptomes—next generation tools for tissue exploration. BioEssays, 42(10), 1900221.
[2].Bergenstråhle, L., He, B., Bergenstråhle, J., Abalo, X., Mirzazadeh, R., Thrane, K., ... & Maaskola, J. (2021). Super-resolved spatial transcriptomics by deep data fusion. Nature Biotechnology, 1-4.
本文由 SEQ.CN 作者:白云 发表,转载请注明来源!